
Compteur de cellules
Au compteur de cellules : Cellules HepG2
Les cellules HepG2 sont une lignée cellulaire provenant du tissu hépatique d’un patient présentant un carcinome hépatocellulaire (CHC). Il est souvent utilisé comme système modèle pour le CHC, ainsi que pour les études sur le métabolisme et la toxicité des médicaments. Les cultures sont adhérentes, avec une morphologie épithéliale, et ont tendance à se développer en petits agrégats qui compliquent la numération des cellules individuelles. Dans les bonnes conditions de culture, elles peuvent former des surfaces apicales et basales distinctes adaptées aux études d'affections hépatiques.
Visionner le webinaire StainFree Cell Detection
Télécharger la note d’application StainFree Cell Detection
Télécharger la note d’application StainFree Cell Detection (Chine)
Télécharger le livre électronique : comptez les cellules comme un-e pro
Figure 1 : Détermination StainFree de la zone couverte en pourcentage

Les cellules HepG2 ont été évaluées par imagerie avec le cytomètre d’imagerie SpectraMax MiniMax 300, et les cellules ont été identifiées à l’aide d’un paramétrage d’analyse personnalisé défini par l’utilisateur. À gauche se trouvent les images d'origine par lumière transmise, et à droite se trouvent les mêmes images avec des masques violets indiquant les cellules identifiées par le logiciel. Deux densités cellulaires sont illustrées pour comparaison.
Figure 2 : Analyse StainFree vs. par fluorescence de la zone couverte en %
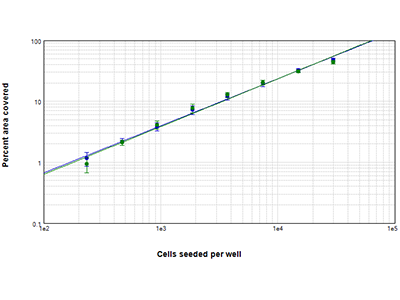
La confluence des cellules HepG2 ensemencées à des densités comprises entre 234 et 30 000 cellules par puits a été analysée avec la technologie StainFree (points bleus), ou les cellules ont été colorées avec le colorant pour essai de cellules vivantes EarlyTox™ et la zone fluorescente a été analysée (points verts). La zone couverte en pourcentage pour une région d’intérêt de l’image pour les deux méthodes correspond étroitement sur l’intégralité de la plage des densités cellulaires.
Figure 3 : Essai EarlyTox Caspase-3/7 NucView 488
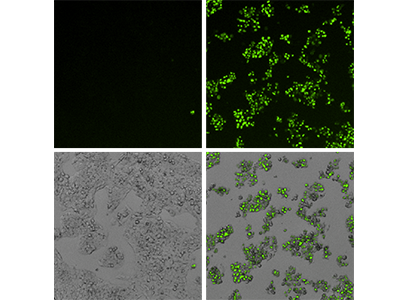
Des cellules HepG2 traitées avec du milieu témoin (panneaux de gauche) ou 200 µm de capsaïcine (panneaux de droite) ont été testées avec le kit de test EarlyTox Caspase-3/7 NucView 488 et imagées avec le cytomètre d’imagerie SpectraMax MiniMax 300 avec les canaux fluorescents verts et de lumière transmise (TL). Ligne du haut : image par fluorescence montrant des cellules apoptotiques marquées par fluorescence verte. Ligne du bas : images par TL et par fluorescence superposées montrant l’apoptose minimale dans les cellules témoins et une apoptose de près de 100 % dans les cellules traitées.
Figure 4 : Test EarlyTox Caspase-3/7 NucView 488 (dans le temps)
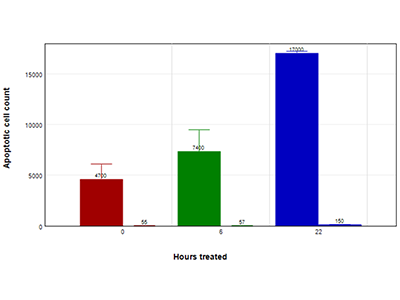
Cellules HepG2 traitées avec 200 µm de capsaïcine (barre de gauche) ou du milieu témoin (barre de droite), pendant 4,5 heures (barres rouges), 6 heures (barres vertes) ou 22 heures (barres bleues). L’apoptose a été évaluée avec le kit d'essai EarlyTox Caspase-3/7 NucView 488. Les cellules ont été incubées avec 5 µm de substrat NucView 488 et imagées aux points indiqués dans le temps.
Figure 5 : Kit d'essai EarlyTox Caspase-3/7 R110 et kit d'essai EarlyTox Glutathione
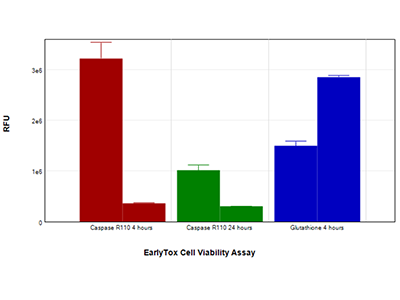
Les cellules HepG2 traitées avec 200 µm de capsaïcine (barre de gauche de chaque paire) ou du milieu témoin (barre de droite de chaque paire) ont été testées avec le kit d'essai EarlyTox Caspase-3/7 R110 (barres vertes et rouges), qui mesure l’activité de la caspase, ou avec le kit d'essai EarlyTox Glutathione (barres bleues), qui mesure la diminution de la glutathione, un indicateur précoce de l’apoptose. Les deux essais ont été détectés sur le lecteur de microplaques multimode SpectraMax i3x en utilisant des protocoles préconfigurés dans le logiciel SoftMax Pro.
Astuce : Les cellules HepG2 se développent en agrégats ou amas de cellules de forme irrégulière qui ne peuvent pas être distinguées à l'œil nu. Une façon utile de quantifier la croissance ou la confluence des cellules HepG2 est d’utiliser la technologie StainFree™ pour déterminer la zone couverte par les cellules. Les images recueillies dans le canal TL peuvent être facilement analysées avec le paramétrage Analyse de champ. Le paramètre d’analyse prédéfini « Cellules » peut être utilisé, ou sinon une analyse personnalisée peut être conçue en utilisant les outils de dessin dans le logiciel SoftMax Pro.
Kit d’outils d’analyse des cellules HepG2
- Plateforme de détection de microplaques multimode SpectraMax® i3
- Cytomètre d’imagerie SpectraMax®MiniMax™ 300
- Logiciel SoftMax ® Pro Software
Paramétrages de l’instrument
Type d’analyse : Analyse de champ
Longueur d’onde pour trouver des objets : TL
À propos de la technologie StainFree Cell Detection
Les essais d’imagerie cellulaires nécessitent généralement l'utilisation de sondes fluorescentes pouvant se révéler toxiques pour les cellules vivantes ou ne peuvent être utilisées que dans des cellules fixées. Une méthode sans marquage pour analyser les numérations cellulaires et la confluence des cellules vous permet de surveiller quantitativement la prolifération et la santé des cellules sans flux de travail fastidieux pouvant perturber la viabilité cellulaire.
La plateforme de microplaques multimode SpectraMax i3 avec le cytomètre d’imagerie MiniMax 300 utilise la technologie unique StainFree Cell Detection en attente de brevet, qui vous permet de réaliser des essais de prolifération cellulaire, de cytotoxicité et d’autres essais sans colorations nucléaires tels que DAPI, qui s’intercale avec l’ADN, et sans colorants de cellules vivantes qui sont toxiques à long terme pour les cellules.