
Logiciel d’automatisation de la microscopie et d’analyse d’images MetaMorph [production arrêtée]
*VEUILLEZ LIRE* Avis d’arrêt de production
MOLECULAR DEVICES évalue régulièrement les technologies d’automatisation de la microscopie et d’analyse d’image afin de s’assurer qu’un produit répond bien aux attentes de ses clients. Après une réflexion approfondie, nous avons décidé de cesser de proposer la suite logicielle MetaMorph, notamment MetaFluor et MetaVue, à partir du 7 juillet 2023. Cette décision s’inscrit dans notre stratégie de fourniture de nouveaux produits innovants avec une sécurité, une conception et une fonctionnalité améliorées en soutien de la recherche scientifique.
Logiciel d’imagerie microscopique avec logiciel d’analyse microscopique en option
Le logiciel d’automatisation de la microscopie et d’analyse d’images MetaMorph® automatise l’acquisition, le contrôle des dispositifs et l’analyse d’images. Il intègre facilement des périphériques et matériels de microscopie à fluorescence différents en un poste de travail unique et personnalisé. Ce logiciel réunit de nombreux modules d’application conviviaux destinés à des analyses spécifiques à la biologie. Deux modules complémentaires, le logiciel MetaFluor® pour l’imagerie de ratio de fluorescence et le logiciel MetaVue® pour l’acquisition et le traitement simples d’images, sont compris dans la gamme.

Prise en charge de microscopes tiers
Le logiciel MetaMorph prend en charge de nombreux microscopes, émetteurs laser et optiques TIRF disponibles sur le marché, et peut être utilisé sur les systèmes d’imagerie déjà installés compatibles.

Mesure de l’imagerie de ratio de fluorescence
Le logiciel d’imagerie de ratio de fluorescence MetaFluor® est conçu pour les mesures d’ions intracellulaires à une ou plusieurs longueurs d’onde, offrant une meilleure compréhension de l’échange d’ions et des fonctions intracellulaires.

Documentation et analyse d'images
Le système d’imagerie de recherche MetaVue™ est un logiciel simple et facile à utiliser permettant d’acquérir et de traiter des images, d’effectuer des fonctions graphiques puis d’archiver et de récupérer des images.
Caractéristiques

Traitement des images en temps réel
Le traitement des images est pris en charge par une accélération du processeur graphique. Cela permet la visualisation d’objets subcellulaires d’à peine 20 nm spatialement et 40 nm axialement.

Module d’acquisition multidimensionnel
Permet la capture de séquences d’acquisition complexes en utilisant une interface utilisateur flexible et guidée.
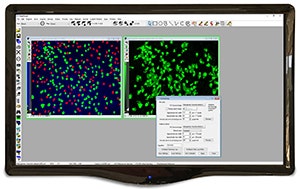
Analyse morphométrique intégrée
Mesure et classe les objets dans des catégories discrètes définies par l’utilisateur, en fonction de toute combinaison de paramètres morphométriques comme, par exemple, la forme, la taille ou la densité optique.

Module de lecture de lames
Automatise l’acquisition d’images multiples et les assemble de manière intégrée. Idéal pour les échantillons tissulaires de grande taille, ce module garantit la reproductibilité tout en éliminant l’approximation des expériences de superposition.

Streaming des appareils et caméras
Accélère le taux de capture des images et transfère simultanément les images vers la mémoire durant l’acquisition, permettant ainsi de capturer des événements cellulaires dynamiques, notamment pour des applications sur cellules vivantes ou d’imagerie cinétique.

4D viewer avec mesures 3D
Outils de visualisation multidimensionnelle, comprenant des empilements d’images séquentielles ainsi que des coupes Z, des longueurs d’onde, des points de capture dans le temps et des positions multiples. Les données peuvent être traitées aux fins de visualisation et de rotation des isosurfaces 3D.
Dernières ressources
Applications particulières
Avancées de nos clients

RÉUSSITES
