Qu’est-ce que l’édition génomique ?
L’édition génomique est une manipulation génétique au cours de laquelle l’ADN génomique d’un organisme vivant est supprimé, inséré, remplacé ou modifié. L’édition génomique est un ciblage spécifique à un site permettant de créer des cassures dans l’ADN par le biais de diverses techniques et ne fait pas toujours intervenir les mécanismes de réparation. Elle consiste en deux techniques : l’inactivation et la correction.
L’inactivation implique la désactivation d’un gène cible, et la correction facilite la réparation du gène défectueux par le biais d’une cassure dans le gène. L’édition génomique présente un vaste potentiel dans de nombreux domaines, dont le développement de médicaments, la chirurgie génique, les modèles animaux, la recherche et le traitement des maladies, l’alimentation, les biocarburants, la synthèse des biomatériaux, etc.
Bien que CRISPR, une technique majeure d’édition génomique, ait été largement utilisée récemment, l’édition génomique a été étudiée pour la première fois à la fin des années 1900. Depuis l’apparition de CRISPR, qui était auparavant une application ambitieuse, la thérapie génique est devenue l’application la plus recherchée de l’édition génomique. Deux approches permettent d’y parvenir : l’ajout de gènes, qui s’additionne au matériel génétique existant pour compenser les gènes défectueux ou manquants, et l’édition génomique, qui traite les maladies en modifiant directement l’ADN lié à la maladie.
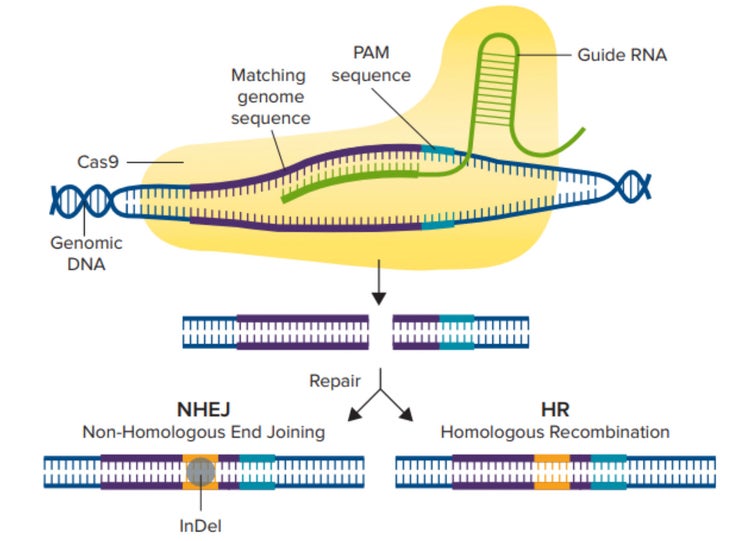
Mécanisme CRISPR/Cas9. L’enzyme Cas9 est activée en se liant d’abord à un ARN guide, puis en se liant à la séquence génomique correspondante qui précède immédiatement la séquence de 3 nucléotides appelée PAM. L’enzyme Cas9 crée alors une cassure double brin, et la voie NHEJ ou HDR est utilisée pour réparer l’ADN, ce qui donne une séquence génétique modifiée.
Un ARN guide (ARNg) similaire à un ARNc est conçu pour cibler une région du gène, et l’enzyme Cas9 peut créer des cassures double brin dans cette région spécifique du génome de la cellule hôte (Figure 1). Après une cassure double brin, la cellule suit l’une des deux voies de réparation suivantes : la voie de la jonction des extrémités non homologues (NHEJ) ou la voie de recombinaison homologue (HDR). La voie NHEJ est généralement utilisée pour perturber les gènes au moyen d’insertions ou de délétions de bases (indels), tandis que la voie HDR peut être utilisée pour introduire un gène rapporteur ou une séquence modifiée en échangeant des séquences entre deux molécules d’ADN similaires ou identiques.
Extrapolation de l’édition génomique grâce à l’ingénierie CRISPR
« CRISPR » : Clustered Regularly Interspaced Short Palindromic Repeats (répétitions palindromiques regroupées et régulièrement espacées). Ces séquences d’ADN ont été découvertes pour la première fois dans le système immunitaire de procaryotes, tels que les bactéries et les archées, et ont gagné en importance en tant qu’outil d’édition génomique depuis 2012 (Jinek et al., 2012). Elles sont très prometteuses dans un grand nombre d’applications, notamment dans l’agriculture, la modélisation des maladies, la thérapie génique et la découverte de médicaments, pour n’en citer que quelques-unes. Sa précision en fait un outil parfait pour l’insertion (knock-in), la suppression (knock-out) et d’autres modifications des séquences d’ADN. Cet outil a remplacé dans une large mesure les outils d’édition génomique existants, fastidieux et coûteux, comme TALENS et ZFNS.
Les séquences CRISPR contiennent de l’ADN d’envahisseurs viraux antérieurs, appelés espaceurs, après chaque répétition palindromique, ce qui facilite la détection et la destruction de futurs virus similaires. La compréhension de ce mécanisme (Jinek et al., 2012) a conduit à la première utilisation de CRISPR dans des cellules eucaryotes (Cong, L, et al., 2013), puis dans d’autres types de cellules et d’organismes appartenant à différents domaines. Le système CRISPR – Cas9 comporte deux composants principaux qui forment un complexe ribonucléoprotéique. Le premier composant ou ARN guide se lie à une séquence d’ADN complémentaire dans le génome et le second composant Cas9 de Streptococcus pyogenes (SpCas9) effectue une cassure double brin au site de la cible. Un motif de reconnaissance du proto-espaceur (PAM) est l’endroit où la nucléase se fixe initialement pour que la coupure en amont ait lieu. Les différentes nucléases CRISPR ont des sites PAM différents et, une fois la coupure effectuée, le système de réparation des cellules est activé et les modifications du génome sont également lancées.
Flux de travail d’édition génomique
Le flux de travail d’édition génomique utilisant les mécanismes CRISPR pour obtenir une lignée cellulaire modifiée confirmée comporte plusieurs étapes. L’optimisation efficace de ces étapes grâce aux outils appropriés permet de réduire le temps, les efforts et les coûts de diverses avancées scientifiques. Cette approche permet d’accélérer la R&D, de révolutionner la découverte de médicaments, la guérison de maladies, la production de cultures génétiquement modifiées, etc. Nous abordons les étapes à suivre et les solutions efficaces que nous proposons pour aider les communautés scientifiques du monde entier à réaliser leurs projets grâce à l’édition génomique.
Solutions de recherche pour valider les modifications des gènes CRISPR/Cas9
La gamme d’instruments de Molecular Devices peut être utilisée efficacement pour réaliser des expériences de criblage, garantissant ainsi le succès des projets d’édition génomique. Avantages du nouvel imageur CloneSelect Imager Florescence (CSI-FL) : assurance de la monoclonalité au Jour 0 dès l’impression des cellules uniques, efficacité de la transfection, confluence des cellules et données de criblage par fluorescence multicanal pour valider l’efficacité de l’édition génomique grâce à des temps de suivi plus courts, un faible risque de plusieurs passages et la robotique.
De plus, notre lecteur de microplaques multimode SpectraMax i3x peut être utilisé pour évaluer l’efficacité de la transfection, surveiller la croissance cellulaire, quantifier l’ADN et les protéines et valider les modifications CRISPR/Cas9 grâce à l’analyse ScanLater Western Blot. Des images haute qualité des autophagosomes peuvent être acquises à l’aide du système confocal ImageXpress Micro, tandis que le logiciel MetaXpress HCI peut identifier et quantifier les autophagosomes individuels de chaque cellule, ce qui nous permet d’analyser les changements phénotypiques résultant des modifications des gènes CRISPR/Cas9.
